シロアリ腸内原生生物の核内には2種の細菌が共生し、
そのゲノム断片は原生生物のゲノムに水平伝播している
2014.4.15
本郷裕一 准教授(生体システム専攻)
◇ポイント◇
- 長年の謎だったシロアリ腸内原生生物の核内共生体の同定に成功
- 核内には2種の真正細菌(Verrucomicrobia門)が同時に共生
- 核内共生細菌2種のゲノム断片が独立に原生生物のゲノムに移行
- 移行した細菌ゲノム断片は原生生物ゲノム内で多数回の重複
生体システム専攻の本郷裕一准教授らは、1944年の発見以来謎であった、シロアリ腸内原生生物の核内に共生する微生物の正体を突き止めることに成功しました。
本郷研究室の桑原宏和研究員、山田明徳研究員(現・長崎大准教授)、木原久美子研究員、修士課程の藤田一麿、独立行政法人理化学研究所・微生物材料開発室(JCM)の大熊盛也室長、野田悟子客員研究員(山梨大准教授)、佐藤朋之博士(現・中外テクノス)らの研究グループによる成果です。
シロアリは木造建築物の大害虫である一方、その強力な木質分解能力は、次世代バイオ燃料開発への応用という観点から、新たな注目を集めています。
シロアリは自身でも木質の一部を分解できますが、大部分は腸内に共生する多様な微生物(原生生物と細菌)が分解・発酵し、さらに空中の窒素を吸収して補ってくれています。
シロアリと腸内微生物はお互いの生存に必須で、絶対的な共生関係にあります。
シロアリ腸内微生物の群集構造は複雑で、微生物同士も強い共生関係を持っています。
例えば腸内原生生物自身の細胞の表面、細胞内さらに核内にも、細菌が共生しています。つまり「マトリョーシカ」のような多重構造になっています。
今回、核内共生体の同定を試み、その結果、腸内原生生物トリコニンファの核内には、ベルコミクロビア門の2種の新種細菌が同時共生していることがわかりました。
さらに、これら2種の細菌のゲノム(全遺伝情報)の一部がトリコニンファの核ゲノム上に移行し、その後、核ゲノム上で多数回、重複していることも明らかになりました。
今後、核内共生体のゲノムが原生生物の核ゲノムに移行後、どのように進化してきたのか、機能に影響を与えているのか、などを研究していきます。本研究の成果は、The ISME Journal (Nature publishing group)にonlineで掲載中です(doi: 10.1038/ismej.2013.222)。
1. 背景
シロアリ腸内には数種類の原生生物(単細胞真核生物:大きさ10–300 µm)と数千種類の細菌(原核生物:大きさ0.5–10 µm)が常に共生している。シロアリが1-2億年前にゴキブリの一種から進化する以前から、それらは祖先ゴキブリの腸内に共生していたと考えられている。他の環境では全く見られない特異な系統群であり、腸内微生物を除去すると、シロアリは死んでしまう。つまり、シロアリとは、腸内原生生物と細菌を含めた複数の生物種からなる「超個体」と言ってもよい。
1億年以上腸内で共存してきた原生生物と細菌の間にも強固な共生関係が構築されており、ほとんどのシロアリ腸内原生生物種は、その細胞表面、細胞質、さらには核内にまで、特定の細菌種を共生させている。核は真核生物のゲノムDNAの収納所であり、生命にとって最も重要な場所であるにもかかわらず、異なる生物種を住まわせるというのは、どういうことであろうか。
シロアリ腸内原生生物の一種であるトリコニンファの核内に「微生物が寄生」していることは、Harold Kirbyによって1944年に初めて報告された。それ以降、様々なシロアリ腸内原生生物種の核内に同様の微生物が存在することが、透過型電子顕微鏡観察によって報告されてきたが、その正体は不明であった。
本研究では、長年の謎であった、シロアリ腸内原生生物の核内共生体の正体を明らかにし、宿主の進化への影響を考察した。
2. 材料と方法
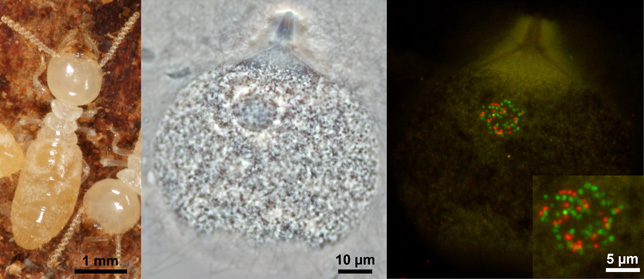
ヤマトシロアリ(Reticulitermes speratus)(図1左)の腸内容物を緩衝液に懸濁し、原生生物トリコニンファ・アギリス(Trichonympha agilis)(図1中)を1細胞だけマイクロマニピュレーターで回収した。界面活性剤でトリコニンファの細胞膜を破壊して核のみを分離し、DNA分解酵素液中で核膜をマイクロメスで切り裂いた。この作業で、原生生物の核DNAは分解され、核内共生体が持つDNAだけを残すことができる。
アルカリ溶液で酵素失活、細菌細胞溶解、DNA変性を同時に行い、Phi29 DNA polymeraseを用いて等温全ゲノム増幅した。これにより、培養を介さずに、単一の原生生物核由来の核内共生体(数十細胞/核)のDNAを数十億倍にコピーし、遺伝子資源として何度も実験に使用できるようにした。
ゲノム増幅産物を鋳型とした遺伝子増幅反応(PCR)により、分類マーカーである16S rRNA遺伝子を特異的に増幅し、塩基配列を解析した。得られた配列のrRNAを特異的に検出する蛍光in situハイブリダイゼーション(FISH)を行い、由来細菌細胞の局在を確認した。また、透過型電子顕微鏡で核内を観察した。
3. 結果と考察
本課題には数年前から取り組んでいたが、細菌の分類マーカーである16S rRNA遺伝子をPCR増幅すると、なぜか通常の同遺伝子よりも短く、かつ高度保存領域にも突然変異を蓄積した偽遺伝子(突然変異により機能を喪失した遺伝子)ばかりが取得されていた。しかも、その16S rRNA偽遺伝子には多数の配列バリエーションが存在した。今回、DNA分解酵素で宿主核のDNAを消化したサンプルを使用したところ、偽遺伝子のPCR産物は消失し、通常サイズの真正な16S rRNA遺伝子配列を初めて取得できた。これは、細菌16S rRNAの偽遺伝子が細菌ゲノム上ではなく、宿主である原生生物の核ゲノム上に存在することを意味している。
得られた配列を分子系統解析したところ、Verrucomicrobia(ベルコミクロビア)門に属する、シロアリ腸に特異的な新規系統群の2種であることがわかった。これら2種をそれぞれ標的としたFISH解析を試みたところ、2種ともにトリコニンファの核内に同時に、あるいはどちらか一方だけが検出された(図1右)。つまり、2種は単独で核内に共生することもあれば、同時に共生することもあり、またどちらも共生していないトリコニンファ細胞もあった。電子顕微鏡像を図2に示した。
これまでに取得した16S rRNA偽遺伝子の分子系統解析を行ったところ、2種の核内共生体それぞれの配列が偽遺伝子化したものであった。さらに分析すると、2種の核内共生細菌のゲノム断片は、それぞれ異なる時期に宿主の核ゲノム上に組み込まれ、その後、それらは何度も宿主核ゲノム内でコピーされ、各コピーが独立に突然変異を蓄積してきたと推定された。真核生物のゲノム上に、細菌由来と考えられる配列が見出されることは珍しくないが、一般に起源は不確かであり、今回のように起源が明白な例は稀である。今後、トリコニンファの核ゲノムの構造を調べることで、共生体からどのようにゲノムが移行し、宿主ゲノム内でどのように進化し、それが宿主および共生細菌自身に影響を与えているのかを、これらのシングルセル・ゲノミクス(単一細胞からのゲノム解析)などにより解明していく予定である。

図1.ヤマトシロアリ(左)と腸内原生生物トリコニンファ(中央、位相差像)、同蛍光像(右)
2種の核内共生細菌(ベルコミクロビア門)が細胞内に持つ16S rRNAを特異的に赤と緑の蛍光でそれぞれ染色して検出した。
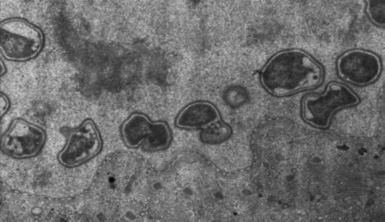 図2. 核内共生細菌の透過型電子顕微鏡像。写真で核膜の内側にあるのが、ベルコミクロビア門細菌である。分裂中のものも見られる。
図2. 核内共生細菌の透過型電子顕微鏡像。写真で核膜の内側にあるのが、ベルコミクロビア門細菌である。分裂中のものも見られる。